크리스퍼 유전자가위에 비해 염기교정, 프라임교정 기술은 20배 낮은 빈도로 발생
[연구필요성]
크리스퍼-카스 시스템을 이용한 유전자 편집 기술은 기초 연구부터 유전질환 치료까지 다양한 분야에 적용된다. 특히 마땅한 치료제가 없는 유전성 희귀 난치 질환에 대해 근본적인 원인이 되는 DNA 돌연변이를 교정해낼 수 있다는 점 때문에 크게 각광받고 있다. 하지만 DNA는 한 번 편집되면 영원히 유지된다는 점에서 보다 높은 정확도와 낮은 부작용이 요구된다. 따라서 현재 개발된 크리스퍼 기술 기반의 유전자 편집 도구들의 안전성을 확인하는 것은 매우 중요한 일이다.
[연구성과/기대효과]
최근, 크리스퍼 유전자가위에 의해 DNA의 이중나선이 절단되고 나면, 예상과 다르게 수백, 수천 개의 긴 DNA 염기서열이 손실되는 결과가 보고된 바 있는데, 아직까지 그 기전은 밝혀지지 않았다. 본 연구에서는 유전자 스크리닝 기법을 통해 긴 DNA 염기서열 손실이 발생하는 현상의 원인이 되는 DNA 수선기작을 밝혀냈다. 또한, 크리스퍼 유전자가위 뿐 아니라, 염기교정, 프라임교정 기술 모두 20배 정도의 낮은 빈도지만, 긴 DNA 염기서열 손실이 발생한다는 것을 밝혀냈다. 이렇게 긴 DNA 염기서열 손실이 발생하는 문제의 원인을 규명함으로써, 향후 보다 안전하고 정확한 유전자 편집을 통해 유전자 치료가 가능할 것으로 기대된다.
[본문]
서울대학교 배상수 교수 (의과대학 의과학과) 연구팀은 서울대학교 김선 교수 (컴퓨터공학부), 서울대학교 김찬혁 교수 (첨단융합학부) 연구팀과 함께 저명한 바이오공학 저널인 Nature Biomedical Engineering 지에 논문을 게재하였다. 이번에 게재된 논문 제목은‘Large DNA deletions occur during DNA repair at 20-fold lower frequency for base editors and prime editors than for Cas9 nucleases’로 크리스퍼 유전자 편집 과정에서 발생할 수 있는 큰 DNA 손실에 관한 연구이다.
크리스퍼-카스9 유전자가위는 DNA 이중가닥 절단을 유도하는데, 이로 인해 예상과 다르게 수백, 수천 개의 긴 DNA 염기서열이 손실된다는 결과가 지속적으로 보고되고 있다. 하지만, 세포 내에서 긴 DNA 염기서열 손실이 발생하는 메커니즘이나 이와 관련된 유전자들이 밝혀져 있지 않았다. 본 연구에서는 약 800여 개 유전자에 대한 유전자 스크리닝 기법을 통해 긴 DNA 염기서열 손실과 관련이 있는 유전자들을 새롭게 발굴하였다. (그림 1. 유전자 스크리닝 기법 설명) 그리고, 이를 통해 긴 DNA 염기서열 손실은 DNA 수선기작들 중 DNA 말단 절제 및 POLQ 중합효소에 의한 수선(TMEJ; Pol theta-mediated end joining)과 매우 크게 연관되어 있다는 것을 처음으로 규명하였다. (그림 2. 실험 결과 설명)
본 연구에서는 더 나아가서 크리스퍼-카스9 유전자가위 외에 염기교정 및 프라임교정 유전자가위에 대해서도 연구를 진행하였다. 염기교정 및 프라임교정 기술은 DNA의 이중나선을 완전히 절단하지 않은 채, 각각 특정 염기를 치환하거나 수십개의 염기서열을 편집할 수 있는 기술이다. 본 연구 결과, 염기교정 및 프라임교정 유전자가위 모두 비록 20배 정도의 낮은 빈도지만, 긴 DNA 염기서열 손실을 마찬가지로 발생시킨다는 것을 처음으로 밝혀냈다.
크리스퍼-카스9 유전자가위를 이용한 최초의 유전자치료제가 지난 2023년 영국과 미국 FDA에서 승인을 받았다. 이제 인간의 DNA를 직접 교정함으로써 유전질환 치료를 할 수 있는 새로운 시대가 열리게 된 것이다. 하지만, 이처럼 크리스퍼 유전자가위의 잠재력이 높은 만큼, 이에 대한 안전성도 보다 면밀하게 살펴보고 검증되어야 한다. 본 연구에서 크리스퍼-카스9 뿐 아니라, 염기교정 및 프라임 교정 유전자가위가 긴 DNA 염기서열 손실을 만들어 내는 과정에 대한 원리를 밝힘으로써 향후 보다 안전하고 정확한 유전자 편집 기술을 만들어 내는데 큰 기여를 할 것으로 기대된다.
이번 연구는 서울대학교 배상수 교수 (의과대학 의과학과) 연구팀이 서울대학교 김선 교수 (컴퓨터공학부), 서울대학교 김찬혁 교수 (첨단융합학부) 연구팀과 함께 공동연구로 진행하였고, 서울대 연구원 황규호 박사, 이석훈 학생이 공동제1저자로 참여하였다.
이번에 게재된 논문은 ‘Large DNA deletions occur during DNA repair at 20-fold lower frequency for base editors and prime editors than for Cas9 nucleases’으로, 세계적 학술지인 ‘Nature Biomedical Engineering (IF=26.8)’지에 11월 5일(한국시간) 게재됐다.
[연구결과]
Large DNA deletions occur during DNA repair at 20-fold lower frequency for base editors and prime editors than for Cas9 nucleases
Gue-Ho Hwang, Seok-Hoon Lee, Minsik Oh, Segi Kim, Omer Habib, Hyeon-Ki Jang, Heon Seok Kim, Youngkuk Kim, Chan Hyuk Kim, Sun Kim & Sangsu Bae
(Nature Biomedical Engineering, https://doi.org/10.1038/s41551-024-01277-5)
[그림설명]
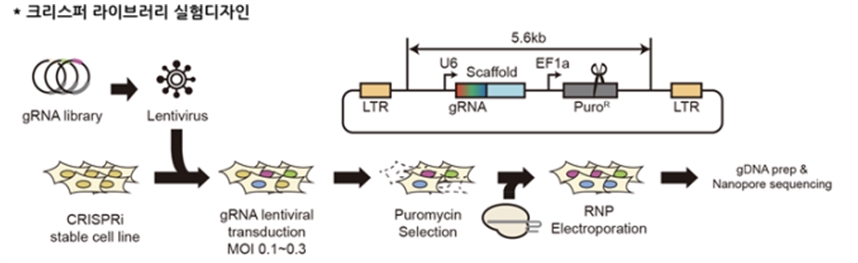
그림 1. 유전자 스크리닝 실험과정 모식도
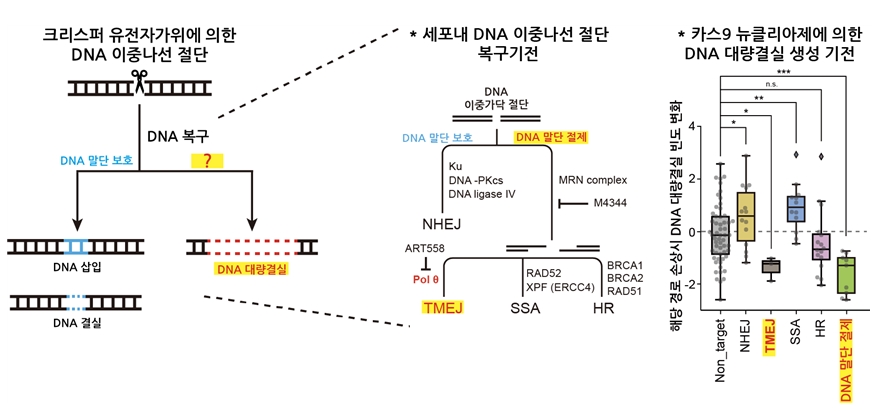
그림 2. 크리스퍼 유전자가위에 의한 이중나선 절단과 이후 DNA 복구과정 모식도. 긴 DNA 염기서열 손실은 DNA 말단 절제 및 POLQ 중합효소에 의한 수선(TMEJ; Pol theta-mediated end joining)과 매우 크게 연관되어 있다는 것을 처음으로 규명함.

