- 슈퍼박테리아의 진화와 출현 분석 -
[연구필요성]
미생물이 갖고 있는 항생제 내성이나 인간에게 유해한 유전자들은 다른 미생물 안으로 쉽게 이동할 수 있어 신/변종 병원균이나 슈퍼박테리아의 출현 원인이 되곤 한다.
이러한 유전자의 이동을 수평적 이동 유전자라고 하며, 이 경로를 파악하기 위해 2015년 HGTree 데이터베이스를 개발하였으나, 이후 유전체 분석 기술의 발전으로 미생물의 유전체 정보량이 급격하게 증가하였고, 이에 따라 많은 양의 유전체의 수평적 유전자 이동 정보를 분석할 수 있는 개선된 분석법과, 이에 따른 데이터베이스의 확장이 필요하였다.
[연구성과/기대효과]
김희발 교수 연구팀이 개발한 HGTree v2.0 데이터베이스는 미생물 유전체 20,536개를 사용하여 총 6,361,199개의 수평적 이동 유전자를 개선된 검출 방법을 통해 확보하고 데이터베이스화하였다.
아울러 수평적 유전자 이동을 탐색할 수 있는 웹 UI를 개선하고 항생제 내성 유전자 정보와 유해 인자의 수평적 유전자 이동 정보를 확인할 수 있도록 하였다.
이번 연구는 국제학술지인 ‘뉴클레익 애시드 리서치 (Nucleic Acids Research, Impact Factor = 19.16)’에 게재되었다. 해당 연구성과는 향후 국내외 미생물의 수평적 유전자 이동 연구에 방향성을 제시할 것이며 이를 바탕으로 세계 각지의 연구진들이 행하는 미생물 연구에 분석 도구로 도움이 될 것이라는 점에서 의미하는 바가 크다.
[본문]
서울대학교 농업생명과학대학 김희발 교수 연구팀은 ㈜eGnome (대표: 조서애)과의 공동연구 결과인 미생물의 수평적 유전자 이동 정보 데이터베이스인 HGTree v2.0를 최근 발표했다.
수평적 유전자 이동 (horizontal gene transfer)은 미생물 개체에서 미생물 개체로 유전형질이 이동되는 현상을 가리키는 유전학 개념이다. 수평적 유전자 이동 현상은 종간의 차이를 뛰어넘어 일어날 수 있으며, 서로 다른 미생물 종의 수평적 유전자 이동을 통해 각 개체는 병원성 유전자 및 항생제 내성 유전자를 전달하고 받을 수 있다.
페니실린의 첫 개발 이후, 150가지 이상의 항생제가 개발되었으며, 이는 인류의 생존에 큰 공헌을 했다. 다양한 항생제의 활용으로 대부분 미생물 사멸하지만, 일부 미생물은 항생제 내성 유전자를 수평적 유전자 이동을 통해 획득하고 생존하여 내성 유전자를 전파하고, 끝내 내성균의 수는 증가하게 된다. 예컨대, 한 병원균이 여러 가지 항생제 저항성 유전자를 수평적 유전자 이동 현상에 의해 획득하면, 어떤 항생제로도 치료할 수 없는 슈퍼박테리아로 진화할 수 있게 되는 것이다 (1번 그림 참조). 2022년 의학학술지 랜싯 (The Lancet)에 발표된 논문에 따르면, 2019년 항생제 내성균에 의한 사망자는 495만 건에 달했으며, 현재는 코로나19로 인한 항생제의 남용으로 슈퍼박테리아가 급증한 것으로 알려져 있다.
인간과 동물의 장내 환경은 수많은 서로 다른 미생물들 사이에서 수시로 수평적 유전자 이동이 일어나는 환경이며, 슈퍼박테리아가 언제든 출현할 수 있다. 이렇듯 미생물의 수평적 유전자 이동의 이해는 인류의 번영에 있어 중요한 주제이지만, 그 중요성에 비해 방대한 분석 계산량 때문에 정밀 분석 데이터베이스 연구는 부재했던 실정이다.
인간에게 큰 위협이 될 슈퍼박테리아의 출현 및 다양한 병원성 유전자, 항생제 내성 유전자의 정보를 분석하고자 2015년 김희발 교수팀은 2,472개의 미생물 유전체를 사용하여 미생물 간의 수평적 유전자 이동을 분석하고 이를 기반으로 HGTree 데이터베이스를 개발했다.
이후 차세대 염기서열 분석법의 발달로 미생물의 유전체 정보가 급격하게 증가했으며, 이에 대한 수평 이동 유전자의 재탐색이 필요했다. 따라서 연구팀은 수평적 이동 유전자 분석 파이프라인을 개선하여 HGTree의 새로운 버전인 HGTree v2.0를 공개했다.
HGTree v2.0는 서열이 완벽하게 구축된 20,536개의 미생물 유전체 사이에서 6,361,199개의 수평적 유전자 이동 (horizontal gene transfer) 정보를 규명하고, 데이터베이스로 공개했다. 이는 현재 발표된 수평적 유전자 이동 데이터베이스 중 가장 많은 데이터를 포함하고 있으며, 수평적 이동이 일어난 유전자의 항생제 내성 및 독성 정보를 예측하여 데이터베이스에 포함했다. 나아가 미생물 유전체 정보를 지속해서 추가하여 가까운 시일에 더 많은 정보를 이용하게 될 것이라고 전해진다.
따라서 본 연구를 통해 공개된 데이터베이스는 미생물의 병원성 유전자 및 항생제 내성 유전자 등의 수평적 이동을 분석하고자 하는 모든 연구자를 위한 데이터베이스이며, 사용자는 HGTree v2.0를 통해 각각의 미생물 유전체 내의 유전정보 및 서로 다른 미생물 종의 유전정보 이동 양상, 미생물 집단 사이에서 발생하는 수평적 유전자 이동 및 유전자 정보를 확인할 수 있다. 또한, 키워드를 통한 특정 유전자 및 해당 유전자의 기능을 검색하고 새로운 미생물 유전체를 사용자가 직접 입력, 데이터베이스 내의 정보와의 수평적 이동 유전자 예측도 가능하다. HGTree v2.0 데이터베이스는 미생물의 분류 및 진화, 나아가 모든 미생물 연구 분야에 분석 도구로써 큰 도움이 될 것으로 기대된다. 해당 연구는 세계적 학술지인 뉴클레익 애시드 리서치(Nucleic acids research)에 11월 09일 온라인 게재되었으며, HGTree v2.0 홈페이지 (http://hgtree2.snu.ac.kr)에서 확인할 수 있다.
본 연구는 한국 연구재단의 4단계 두뇌한국21 프로그램과 농림 식품기술기획평가원 고부가가치 식품기술개발사업의 지원을 받아 진행되었다.
[연구결과]
HGTree v2.0: a comprehensive database update for horizontal gene transfer (HGT) events detected by the tree-reconciliation method
Youngseok Choi, Sojin Ahn, Myeongkyu Park, SaetByeol Lee, Seoae Cho, Heebal Kim
https://doi.org/10.1093/nar/gkac929
2015년에 개발된 HGTree의 새로운 버전인 HGTree v2.0는 기존 데이터베이스보다 약 8배 많은 데이터를 바탕으로 총 6,361,199개의 수평 이동 유전자 정보를 가지고 있다.
[용어설명]
- ○미생물 개체에서 미생물 개체로 유전 형질이 이동되는 현상
[그림설명]
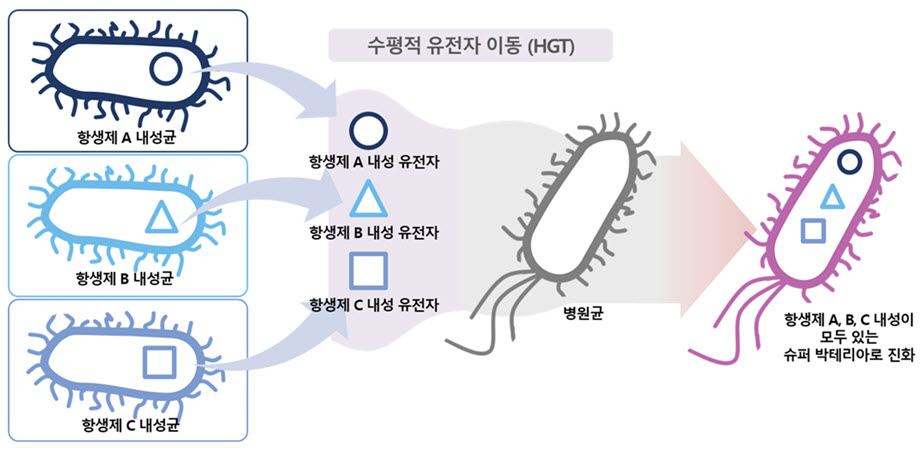
수평적 유전자 이동에 의한 박테리아의 진화 과정. 예를 들어, 항생제 A 내성균, B 내성균, C 내성균으로부터 A, B, C의 세 가지 내성 유전자를 수평적 이동으로 획득한 병원균은 항생제 A, B, C에 모두 내성이 있는 슈퍼박테리아로 진화하게 된다.
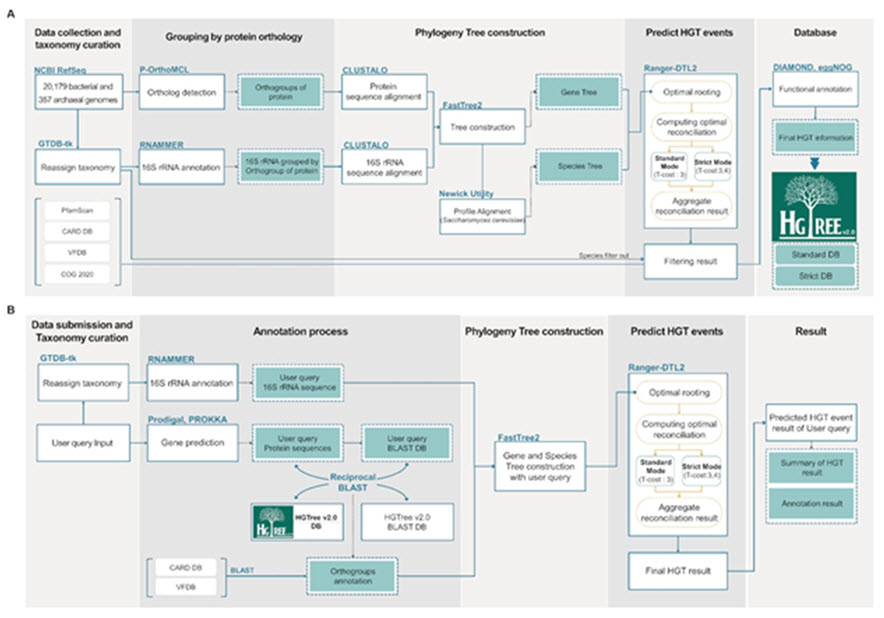
데이터베이스 구축 알고리즘 및 사용자 입력 수평적 이동 유전자 분석 알고리즘. 해당 알고리즘들은 20,179개의 박테리아 유전체와 357개의 고세균 유전체, 총 20,536개의 미생물 유전체를 바탕으로 진행된다.
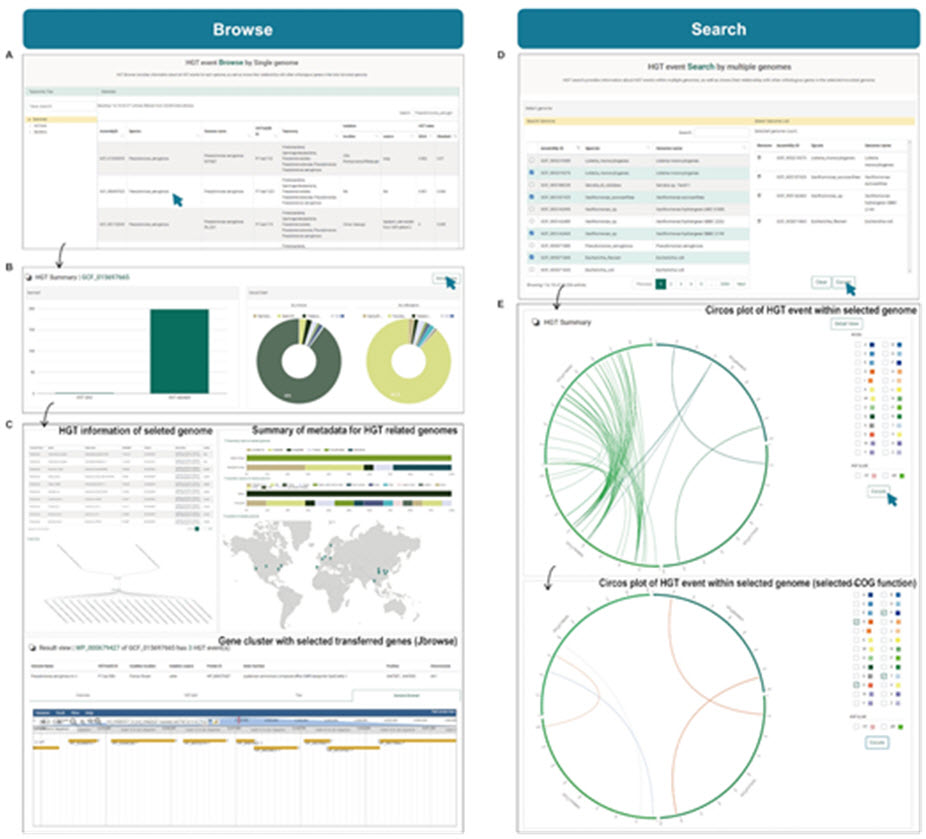
데이터베이스의 여러 가지 시각화 도구들과 그 사용 예시. 사용자는 HGTree v2.0 홈페이지 (http://hgtree2.snu.ac.kr)에서 각각의 미생물 유전체 내의 유전정보 및 유전정보 이동 양상, 미생물 집단 사이에서 발생하는 수평적 유전자 이동 및 유전자 정보를 확인할 수 있다. HGTree v2.0에서 제공하는 여러 가지의 시각화 도구를 통해 사용자는 더 직감적으로 해당 정보들을 접할 수 있다.

