- Genome-wide study on 72,298 individuals in Korean biobank data for 76 traits -
[연구필요성]
영국 바이오뱅크인 UK Biobank와 같은 대규모 바이오뱅크는 수많은 표현형에 대해 유전체 연관성 발견을 가능케 하였고, 복잡한 질병의 유전적 요소를 파악하는데 큰 기여를 하였다. 하지만 이러한 연구는 주로 유럽인들을 대상으로 하며, 한국인을 비롯한 동아시아인에 대한 연구는 충분하게 이뤄지지 않은 상태이다. 유럽인들에게는 거의 존재하지 않는 변이들이 다른 인종에서는 흔하게 존재할 수 있으므로, 다양한 인종에 대한 연구는 유럽인에 대해서는 발견되지 않았던 유전적 요인들을 발견할 수 있도록 할 수 있다.
[연구성과/기대효과]
본 연구에서는 한국의 바이오뱅크인 KoGES의 72,000여명의 유전체 및 표현형 데이터를 통해 76개의 표현형에 대한 전장 유전체 연관성 분석을 수행하였다. 한국인을 대상으로 다수의 표현형에 대한 유전체 연관성 분석을 수행한 것은 최초이며, 이 결과 122개의 유전자 표현형 연관성을 새로이 발견하였다. 또한, 일본의 바이오뱅크인 Biobank Japan과의 메타 분석을 통해 유전 위험 점수 (Polygenic risk score) 모형의 질병 예측 성능이 향상되는 것을 확인하였다. 이는 동아시아인의 유전자와 질병의 연관성을 발견하는 데에 공헌하게 될 것이다.
[본문]
UK Biobank나 FinnGen과 같은 대규모 바이오뱅크는 수많은 표현형에 대해 수십만명 이상의 데이터를 이용하여 전장 유전체 연관성 분석(Genome-wide association study, GWAS)을 가능하게 함에 따라 복잡한 질병의 유전적 요소를 파악하는 데에 큰 기여를 하였고, 이러한 분석은 유전체 기반의 질병 예측 모형으로 이어져 유전적으로 질병 위험도가 높은 사람을 발견하는 데에도 큰 역할을 한다. 이러한 성공에도 불구하고, 현존하는 GWAS는 대부분 유럽인들을 대상으로 분석된 결과이며 비유럽인에 대한 연구는 충분하게 이루어지지 않은 상태이다. 유럽인들에게는 희귀한 변이들이 다른 인종에게는 흔한 변이일 수도 있으므로, 비유럽인에 대한 불충분한 연구는 다양한 인종에 대한 질병의 위험도 예측 등의 추가적인 발견을 저해하는 요소가 된다.
본 연구는 한국의 바이오뱅크 데이터인 KoGES의 72,298명의 유전체 데이터를 이용하여 76개 표현형에 대한 GWAS를 수행하였다. 최근 Biobank Japan (일본), Taiwan Biobank (대만) 등의 대규모 바이오뱅크에서 다수의 표현형에 대한 GWAS를 수행하는 등 비유럽인에 대한 연구가 활발하게 전개되고 있으며, 본 연구의 결과도 동아시아인에 대한 분석 표본 수 증가로 더 많은 유전적 발견을 하는 데에 기여할 것이다. 본 연구는 GWAS에 사용되는 최신 분석 방법을 적용하여 122개의 새로운 연관성을 발견하였으며 다면발현 분석(pleiotropy analysis)을 통해 여러 형질에 영향을 주는 주요 유전자들을 규명하였다. 특히 새로이 발견된 연관성 중 상당수는 유럽인들에게 희귀한 변이였으며, 이는 다양한 인종에 대한 분석의 필요성을 뒷받침해준다.
또한, 동아시아인의 유전적 특성을 확인하기 위해 32개 표현형에 대해 Biobank Japan과의 메타 분석을 수행하였으며, 이를 통해 379개의 새로운 유전자 표현형 연관성을 발견하였다. 이 중 상당수는 개별 바이오뱅크의 분석 결과에서는 발견되지 않았던 것으로, 메타 분석을 통해 연관성 분석의 검정력을 높일 수 있다는 점을 확인하였다. 메타 분석 결과를 이용하여 유전 위험 점수 모형을 구축하여 질병 위험도를 예측한 결과 Biobank Japan의 결과만을 이용하여 구축한 모형에 비해 예측력이 향상되는 것을 확인할 수 있었다. 또한, 다양한 인종에 대한 유전체 연구 활성화를 위해 본 연구의 결과를 누구나 활용할 수 있게 공개하였다.
요약하면, 본 연구는 한국인 데이터의 대량 유전체 분석을 통해서, 한국인 더 나아가서는 동아시아인에 있는 질병의 유전적 요인을 찾고, 이를 바탕으로 새로운 예방 및 치료법을 찾는데 공헌하게 될 것이다.
본 연구는 남기성 박사과정생 주도로 연구재단 해외우수과학자유치사업 (Brainpool+) 지원을 받아 수행되었으며, 유전체 연구 분야의 최상위 저널이자 Cell 의 자매학술지인 Cell Genomics 에 2022년 10월에 게재되었다. 본 연구 그룹은 전세계 바이오뱅크를 통합적으로 분석하기 위한 컨소시움인 Global Biobank Meta-Analysis Initiative (GBMI) 에 참여하고 있으며, 본 연구도 GBMI와 함께 수행되었다.
[연구결과]
Genome-wide study on 72,298 individuals in Korean biobank data for 76 traits
Kisung Nam, Jangho Kim, Seunggeun Lee
Cell Genomics, in press
DOI:https://doi.org/10.1016/j.xgen.2022.100189
Genome-wide association studies (GWAS) on diverse ancestry groups are lacking, resulting in deficits of genetic discoveries and polygenic scores. We conducted GWAS for 76 phenotypes in Korean biobank data, namely Korean Genome and Epidemiology Study (KoGES, n=72,298). Our analysis discovered 2,242 associated loci, including 122 novel associations, many of which were replicated in Biobank Japan (BBJ) GWAS. We also applied several up-to-date methods for genetic association tests to increase the power, discovering additional associations that are not identified in simple case-control GWAS. We evaluated genetic pleiotropy to investigate genes associated with multiple traits. Following meta-analysis of 32 phenotypes between KoGES and Biobank Japan (BBJ), we further identified 379 novel associations and demonstrated the improved predictive performance of polygenic risk scores by using the meta-analysis results. The summary statistics of 76 KoGES GWAS phenotypes are publicly available, contributing to a better comprehension of the genetic architecture of the East Asian population.
[용어설명]
- ○분석하고자 하는 형질 (표현형)과 연관성이 있는 유전자의 위치를 유전체 전반에 걸쳐 확인하고 탐지하는 분석 방법
- ○개별적인 연구들의 결과를 종합하여 합성하는 통계적 방법
[그림설명]
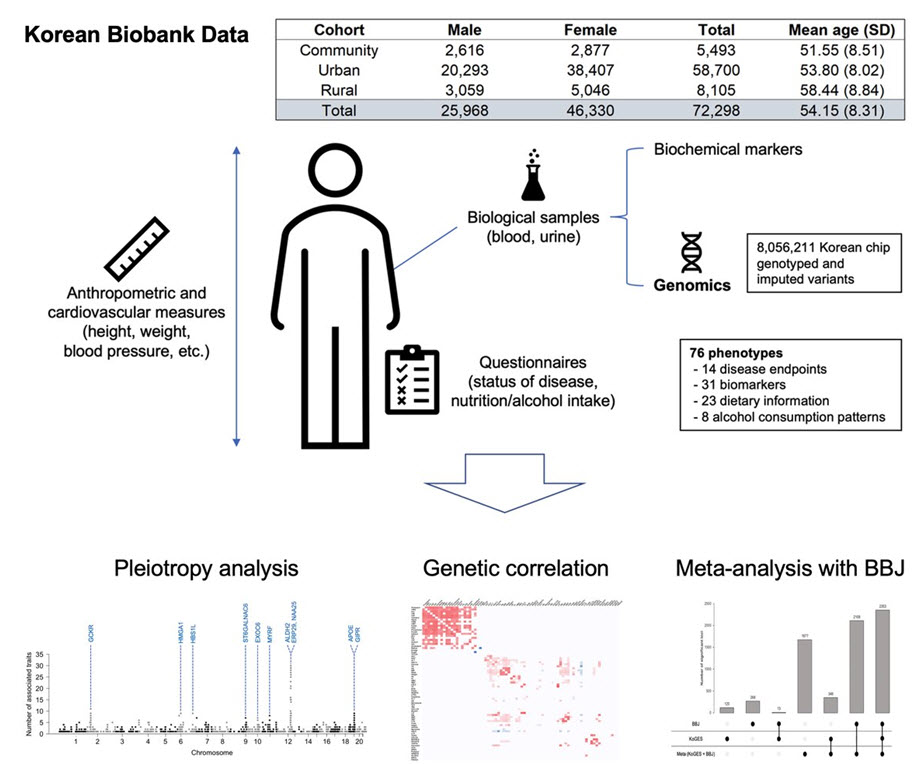
연구 개요

