- 유전체 분야 권위지 Genome Biology에 논문 2편 동시 게재 -
[연구필요성]
서울대학교 김희발 교수팀은 2018년 공식 출범한 척추동물 유전체 프로젝트(Vertebrate genome project, 이하 ‘VGP’)의 리더인 에릭자비스 교수(록펠러 대학)와 척추동물의 표준유전체 구축과 관련하여 긴밀한 국제 공동연구를 수행해 왔다. - 표준유전체 자료는 향후 모든 생물학 연구의 기반이 되므로 표준유전체는 그 자체로 무결해야 한다는 것을 연구팀은 관련 학계에 강조해왔으나, 표준유전체 전반에 포함된 오류의 양과 유형을 계측한 연구는 찾아보기 힘들었다.
서울대학교 김희발 교수팀은 VGP와의 협업 수행을 통해 기존에 조립된 표준유전체로부터 수천개의 유전자 조립 오류를 발견하여 유전체 분야 권위지인 지놈바이올로지(Impact factor 17.91)에 이례적으로 관련 논문 2편을 동시 온라인 게재(9월 27일) 하였다.
[연구성과/기대효과]
이 연구성과는 향후 국내외 표준유전체 구축의 방향성을 제시한다는 점에서 중요하며 이를 바탕으로 국내 교수팀이 국제 표준유전체 구축 분야를 선도할 수 있는 계기를 마련했다는 점에서 의미하는 바가 크다.
[본문]
서울대학교 김희발 교수팀은 국제 공동연구인 척추동물 유전체 프로젝트(Vertebrate genome project, 이하 VGP)와의 협업 수행을 통해 기존에 조립된 표준유전체로부터 수천개의 유전자 조립 오류를 발견하여 유전체 분야 권위지인 지놈바이올로지(Impact factor 17.91)에 이례적으로 관련 논문 2편을 동시 온라인 게재(9월 27일) 하였다.
GP는 지구상에 서식하는 66,000여 종의 척추동물 표준유전체 구축 및 유용 유전자 발굴을 목적으로 하는 전 세계적 프로젝트로 인간과 동물의 질병 연구는 물론 생물의 진화와 멸종위기 종 보전 연구에 적용 가능한 유전체를 구축하는 것이 목적이다. 서울대학교 김희발 교수팀은 2018년 공식 출범한 VGP의 리더인 에릭자비스 교수(록펠러 대학)와 척추동물의 표준유전체 구축과 관련하여 긴밀한 국제 공동연구를 수행해 왔다. 특히 이들은 과거에 구축된 표준유전체와 새롭게 구축된 VGP 그룹의 표준유전체를 정량적으로 비교하는 체계를 구축하였으며 이를 통해 기존의 표준유전체가 포함하는 오류의 양과 유형을 정밀하게 계측하였다. 표준유전체 자료는 향후 모든 생물학 연구의 기반이 되므로 표준유전체는 그 자체로 무결해야 한다는 것을 연구팀은 관련 학계에 강조해왔다. 이번에 새롭게 구축한 표준유전체 비교체계를 이용하여 생물학 분야의 모델 생물로 잘 알려진 금화조, 벌새, 오리너구리, 등목어의 기존 표준유전체에서 각 종별 수백에서 수천개에 달하는 유전자의 결손 및 복제가 유전체 구축 과정 중에 발생한 오류였다는 사실을 발견하였다. 이러한 결과는 기존의 과학적 통념을 넘어서는 많은 양의 유전자 조립 오류를 포함하는 것이다. 만약 이렇게 많은 양의 오류를 포함한 유전체를 기반으로 연구를 수행할 경우 연구자들이 잘못된 결론에 도달할 것은 자명한 것이다. 실제 이번 연구를 통해 과거 학계에 알려진 몇몇 금화조, 벌새, 오리너구리의 종 특이적 진화 특성 조차 유전체 조립과정에서 발생한 오류에 기인하였다는 것을 밝혀내었다.
향후 진화학, 의학, 뇌과학 분야 등 생물학의 많은 부분이 유전체 빅데이터를 바탕으로 연구 되어질 것이며 이들 연구의 기반이 되는 표준유전체의 중요성은 점점 부각될 것이다. 따라서 기존의 표준유전체에 만연해 있는 오류를 선제적으로 발견한 연구 성과는 향후 국내외 표준유전체 구축의 방향성을 제시한다는 점에서 중요하며 이를 바탕으로 국내 교수팀이 국제 표준유전체 구축 분야를 선도할 수 있는 계기를 마련했다는 점에서 의미하는 바가 크다.
[연구결과]
Widespread false gene gains caused by duplication errors in genome assemblies
Byung June Ko, Chul Lee, Juwan Kim, Arang Rhie, DongAhn Yoo, Kerstin Howe, Jonathan Wood, Seoae Cho, Samara Brown, Giulio Formenti, Erich D. Jarvis, Heebal Kim
doi: https://doi.org/10.1101/2021.04.09.438957
기존에 구축된 금화조, 벌새, 오리너구리의 표준유전체 내 수백에서 수천개 유전자의 복제가 유전체 조립오류에 의해 발생한 것을 확인하였다. 이들 유전자 복제 오류의 양과 유형을 계측하였으며 향후 유전자 복제 오류를 방지하기 위해 유전체 조립 시 반수체 구분 및 염기서열 오류 방지의 중요성을 강조하였다.
False gene and chromosome losses in genome assemblies caused by GC content variation and repeats
Juwan Kim, Chul Lee, Byung June Ko, Dong Ahn Yoo, Sohyoung Won, Adam M. Phillippy, Olivier Fedrigo, Guojie Zhang, Kerstin Howe, Jonathan Wood, Richard Durbin, Giulio Formenti, Samara Brown, Lindsey Cantin, Claudio V. Mello, Seoae Cho, Arang Rhie, Heebal Kim & Erich D. Jarvis
doi: https://doi.org/10.1186/s13059-022-02765-0
기존에 구축된 금화조, 벌새, 오리너구리, 등목어의 표준유전체 내 수천개의 유전자가 조립오류로부터 영향을 받아 전체 또는 일부가 손실된 것으로 나타났다. 특히 전체 유전자중 절반 이상의 유전자 프로모터 지역에서 손실오류가 발견되었으며 이는 높은 GC content에 의한 염기서열 해독과정의 편향으로 부터 발생한 것으로 나타났다. 이 결과는 유전체의 조립과정에서 발생한 오류가 잘못된 생물학적 해석을 야기 할 수 있다는 것을 시사한다.
[그림설명]
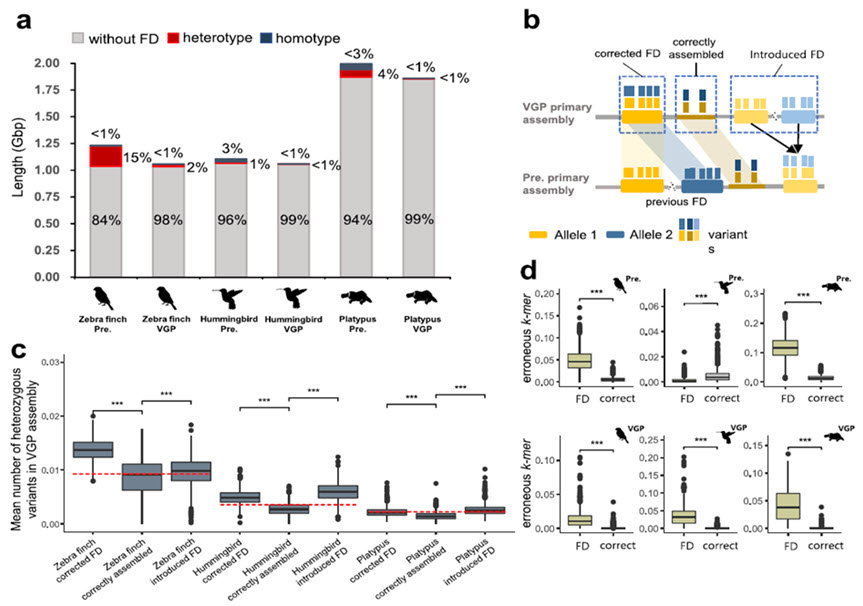
금화조, 벌새, 오리너구리의 유전자 조립 오류에 의한 허위 염기서열 복제 양을 계측하였으며, 이들 복제오류가 높은 이형접합성 및 염기서열 해독 오류와 관련이 있다는 것을 나타낸다.
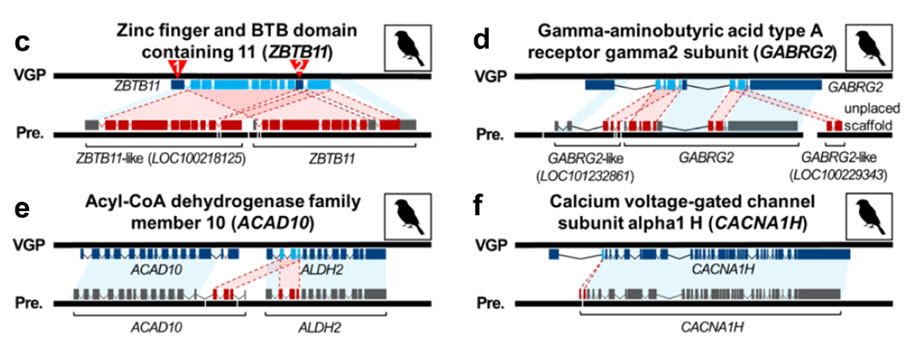
금화조 내 허위 유전자 획득의 네가지 사례. 위 사례는 유전자 전체 혹은 일부가 복제 오류에 의해 새로운 유전자로 잘못 인식된 경우, 각기 다른 유전자가 하나의 유전자에 혼합된 경우, 하나의 유전자 내 엑손 영역이 허위 복제된 경우를 나타낸다.
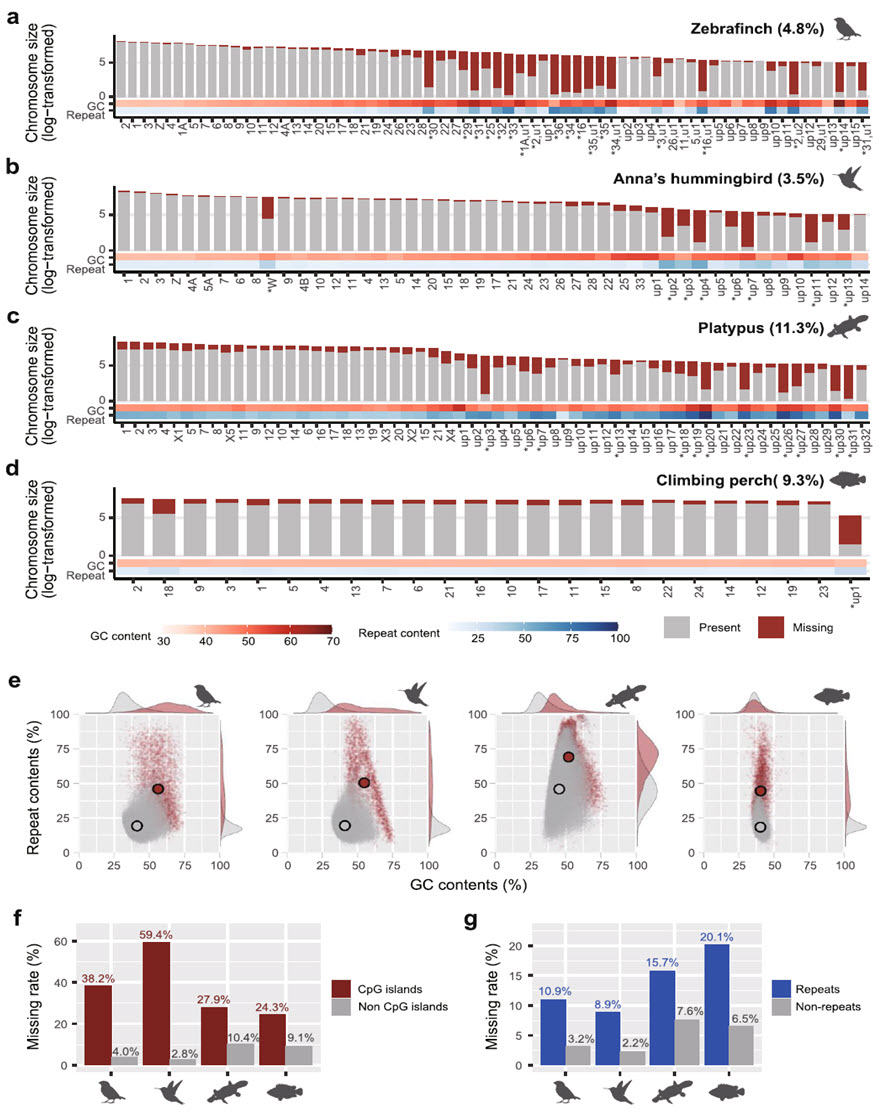
금화조, 벌새, 오리너구리, 등목어의 유전자 조립 오류에 의한 허위 유전자 손실 양을 계측하였으며, 이들 손실 오류가 GC 및 repeat content와 연관이 있다는 것을 나타낸다.
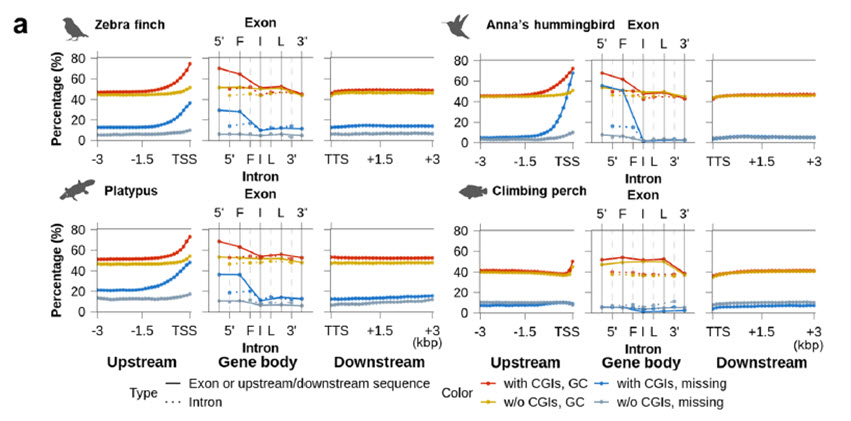
금화조, 벌새, 오리너구리, 등목어의 표준유전체 유전자들의 5말단 지역에서 허위 손실 오류가 빈번히 발생한 것을 밝혀냈다.
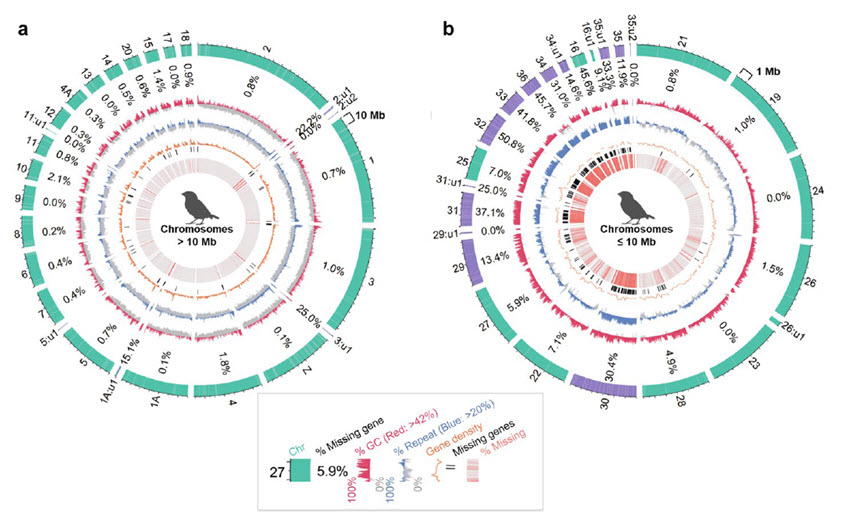
금화조 표준유전체 전반에 걸쳐 허위 유전자 손실 오류를 계측하였으며 이를 통해 금화조의 micro chromosome들을 새롭게 발견하였다.

